
概述
NEBNext DirectTM Cancer HotSpot Panel采用NEBNext Direct 靶向富集方法,能够捕获 50 个基因中 190 个常见癌症目标序列的双链 DNA,能够涵盖 40 kb 的序列,包括18,000 个COSMIC 数据库中所述情况。适用于构建约 150 bp 的文库,适用于Illumina 平台 PE75 测序。
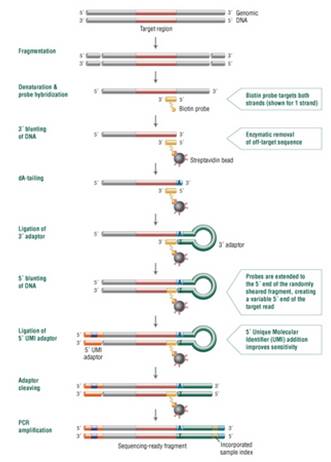
产品原理
在 NEBNext Direct 靶向富集方法中,片段化的 DNA 迅速杂交到生物素化的寡聚核苷酸饵上,该寡聚核苷酸定义了目标序列 3’ 端序列。核苷酸饵-目标序列杂交体结合到链霉亲和素磁珠上,而任何 3’ 脱靶序列则通过酶学法去除。短杂交时间搭配酶学法去除 3’ 脱靶序列的方法与传统的基于杂交的富集方法相比,有着更高的测序效率。
这些经过修饰的目标序列随后被转化为含有唯一分子标签(UMI)及样本 barcode 的文库,与 Illumina 平台兼容。可用于上机测序的文库可以在一天之内制备完成。整个制备流程与大多数自动化仪器设备兼容。
产品优势
1. 靶向富集,配合 NGS 技术,使对基因组中目标区域高通量、高深度的测序成为可能;
2. NEBNext Direct 是一种全新的,基于杂交的捕获方法,与传统的液相杂交及多重 PCR 方法相比有明显的优势;
3. 可比对到目标区域的reads 比例更高;
4. 避免过度测序,减少每个样本的花费;
5. 所有区域均可得到均一的测序结果,无论 GC 含量多少;
6. 将富集和文库制备流程相结合,1 天即可完成;
7. 能够从有限及降解的 DNA 样品中得到高质量的文库,包括 FFPE 及 ctDNA;
8. 区分分子重复,降低假阳性突变并提高敏感度。
NEBNext Direct Cancer HotSpot Panel 能够获得很高的可比对到目标区域序列百分比,即使是对难以建库的样本类型
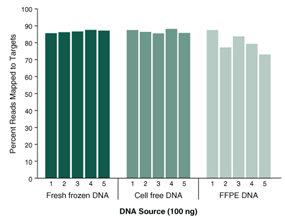
图中显示能够比对到目标序列 Reads 的比例。
每个文库用 100 ng DNA 构建。
Reads 通过 Illumina® MiSeq® 平台测得,使用 2x75 bp 的测序策略,8 bp 的样本 ID 和 12 bp 的唯一分子ID(UMI)。
序列比对通过 BWA-MEM 方法,PCR 重复序列通过唯一分子 ID (UMI)去除。
NEBNext Direct Cancer HotSpot Panel 在所有目标区域有高度均一的覆盖度
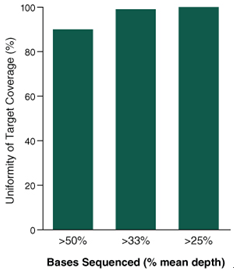
图中显示能够达到平均测序深度50%,33% 及 25% 的目标序列比例。
每个文库用 100 ng DNA 构建。
Reads 通过 Illumina® MiSeq® 平台测得,使用 2x75 bp 的测序策略,8 bp 的样本 ID 和 12 bp 的唯一分子ID(UMI)。
序列比对通过 BWA-MEM 方法,PCR 重复序列通过唯一分子 ID (UMI)去除
NEBNext Direct Cancer HotSpot Panel 偏嗜性最小
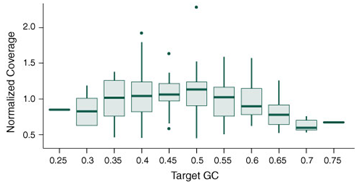
图中显示不同 GC 含量的目标序列归一化后的覆盖度。
每个文库用 100 ng DNA 构建。
Reads 通过 Illumina® MiSeq® 平台测得,使用 2x75 bp 的测序策略,8 bp 的样本 ID 和 12 bp 的唯一分子ID(UMI)。
序列比对通过 BWA-MEM 方法,PCR 重复序列通过唯一分子 ID (UMI)去除。
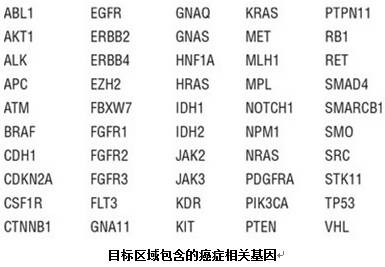
产品信息:

详情请垂询NEB一级代理商北京友谊中联
010-64892983/4
上一篇:没有了