
传统的克隆实验包括以下几种操作:将DNA 片段插入到质粒中,以修饰或引入抗性基因、启动子和信号序列或开放阅读框
等,用于基因表达或下游蛋白表达研究。在开始克隆实验之前定出克隆策略是很重要的。需要考虑的问题有:酶切后是否有
与目标载体匹配的末端,插入序列在载体上可能的方向,以及插入序列是否在读码框内,是否会影响翻译。以下指南有助于
克隆的设计和相关问题的解决。
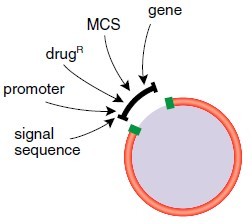
准备插入片段和质粒
来源于质粒的插入片段
• 选择适当的限制性内切酶切割质粒,获得能直接克隆于载体的 DNA 片段。选择产生不同末端的两种限制性内
切酶来进行单向克隆。关于优化限制性内切酶反应的指导和问题解决建议,见 2013﹒2014 商品目录 275-
277 页或 NEB网站的技术参考部分。
来源于 PCR 的插入片段
• 引物设计时引入能单向克隆到载体的限制位点
• 大多数限制性内切酶的识别位点上游需要 6 个保护碱基
• 如需高保真扩增可以选择具有校读功能的聚合酶,如:Q5® 超保真DNA 聚合酶(NEB #M0491)
• PCR 优化指南和问题解决见 2013﹒2014 商品目录 325-326 页或 NEB 网站上技术参考部分
• PCR 产物可通过凝胶电泳回收或离心柱纯化
• 选择用适当的限制性内切酶消化
来源于退火寡核苷酸的插入片段
• 退火的寡核苷酸用来引入 DNA 片段(如启动子和聚合接头等)
• 两条带有互补的 5´ 或 3´ 突出末端寡核苷酸退火后能被连接到用适合的限制性内切酶切割的载体中
• 未磷酸化的寡核苷酸用 T4 PNK (NEB #M0201)
常规退火反应
寡核苷酸 5 µl (10 µM 储液)
10X 连接缓冲液 5 µl
总体积 至 50 µl
温度 85℃ 10 分钟,缓慢降温(30-60 分钟)
载体
• 选择适当的限制性内切酶消化载体,最好使用能产生不匹配末端的内切酶,以防载体自连。
去磷酸化
• 去磷酸化反应是用来防止自身连接。NEB 提供两种 DNA 去磷酸化的产品。
– 小牛肠碱性磷酸酶(CIP)(NEB #M0290)是一种活力较强的去磷酸化酶,在许多不同条件下及各种
NEBuffer 中均有活性。然而,CIP 不能被热失活,因此需要在连接前纯化 DNA。建议 CIP 的使用量不要
超过推荐用量,以达到更好的纯化效果。
– 热敏磷酸酶(AnP)(NEB #M0289)不仅具备 CIP 的所有功能而且还能被热失活。AnP 需要锌离子发挥活
性
AnP 去磷酸化
AnP 1 μl(5 单位)
DNA 1-5 μg
10X 缓冲液 2 μl(终浓度 1 mM)
总体积 至 20 μl
平齐化/末端修复
• 有时插入的片段和载体需要进行末端平齐化/末端修复
• 用具有校读活性的聚合酶进行 PCR,其产物的绝大部分是平末端
• T4 DNA 聚合酶(NEB #M0203)或 Klenow(NEB #M0210)能补平 5´ 突出末端(如 EcoRI-HF),切割
3´ 突出末端(如 PstI-HF)
• 快速末端平齐试剂盒(NEB #E1201)能在 30 分钟内完成 DNA 末端的平齐化和磷酸化
磷酸化
• 连接两个 DNA 片段时,至少其中一个片段的末端应含有 5´ 磷酸(插入片段或载体)
• 引物通常以非磷酸化形式提供,因此 PCR 产物的 5´ 末端为非磷酸化,限制性内切酶消化的 DNA 其 5´ 末端
都有磷酸化
• DNA 片段可以通过与 T4 PNK NEB #M0201)温育进行磷酸化
T4 PNK 磷酸化
T4 PNK 1 μl(10 单位)
DNA 1–2 µg
10X T4 PNK 缓冲液 5 μl
10 mM ATP 5 μl(终浓度 1 mM)
无核酸酶污染的水 至 50 μl
温育 37℃ 30 分钟
纯化载体和插入片段
• 连接反应前,用凝胶电泳切胶回收或离心柱回收方法纯化载体或插入片段
• β-琼脂糖酶 I(NEB #M0392)可以用于纯化低熔点琼脂糖中的 DNA,DNA 的纯化还可用离心柱及树脂法
• 用长波 UV(360 nm)观察琼脂糖凝胶电泳结果,以减少 UV 暴露造成的 DNA 损伤
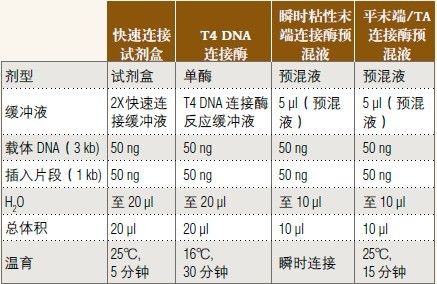
连接载体和插入片段
• NEB 目前提供预混液形式的连接酶,请在 2013﹒2014 商品目录第 100 页的连接酶选择表格中选择
• 载体和插入片段的摩尔比为 1:3
• 室温解冻并混匀连接缓冲液
• 平末端连接需要延长连接时间或使用高浓度的连接酶/连接酶预混液
• 连接后置于冰上并进行转化反应
• 用快速连接缓冲液或连接酶预混液时勿热失活,以免抑制转化反应
转化
• 关于感受态细胞的信息和属性,请参考2013﹒2014 商品目录第 202-213 页
• 提高感受态细胞效率的方法,请参考 2013﹒2014 商品目录第 323 页
• 通过电击的方式转化,建议您选择电转连接酶TM
用 NEB 连接酶连接
使用 Gibson 组装试剂盒克隆
Gibson 组装试剂盒中,包含 NEB 5-alpha E. coli 感受态细胞,经过优化,该试剂盒在一个试管中经等温反
应,可以高效的将 1 或 2 个组装片段插入到任何载体,完成组装和克隆。更多详细信息请参考 2013﹒2014
商品目录第 127 页或登陆 NEB 网站查询。
设计引物
• 为了高效组装 PCR 片段到载体中,建议使用 15-25 个核苷酸重叠序列的引物,且其 Tm 值等于或高于 48℃
• 为了更有效的设计 Gibson 组装引物,建议您登陆 NEBGibson.com 使用在线引物设计软件 NEBuilder 设计
引物,也可以参考 Gibson 组装引物设计说明 PCR 扩增片段
• 建议使用 Q5 超保真 DNA 聚合酶(NEB #M0491)或相关产品(NEB #M0493, NEB #M0492)扩增组装片段
• 以质粒为模板进行扩增时,建议每50μl PCR 反应体系中使用微量的 DNA(0.1–0.5 ng)进行扩增
• 通过凝胶电泳验证 PCR 产物的纯度和产量
• 如果产物纯度 > 90%,就无需对 PCR 产物进行纯化
• 在 Gibson 组装反应体系中,未纯化的 PCR 产物可以占到 20%
• 如果必须使用大量的质粒作为模板,扩增后用 DpnI(#R0176)消化 PCR 产物以降低背景
载体线性化
• 使用限制性内切酶线性化质粒时,NEB 建议使用两种限制性内切酶进行双酶切以降低未切割载体的背景
• NEB 推荐您使用 Q5 超保真 DNA 聚合酶(NEB #M0491)或者相关产品(NEB #M0493,NEB #M0494)扩
增载体
建议反应体系 & 条件
• 纯化用于组装的 DNA,可以溶于 dH2O(推荐使用 Milli-Q 水或同等纯度的水)、TE 或者其它稀释缓冲液
• 当组装 1-2 个片段到一个载体中时,NEB 推荐您使用总量为 0.02-0.5 pmols 的 DNA 片段
• 为了优化组装,在加入 Gibson 组装预混液之前调整 DNA 的体积到 10 μl
• 若 DNA 的体积大于 10 μl,相应的调整 Gibson 组装预混液的体积
• Gibson 组装试剂盒经优化可在 50℃ 15 分钟完成组装。反应时间无需超过 15 分钟,某些情况下延长时间
(60 分钟)可提高效率
• 不要过夜反应
Gibson 组装克隆方法流程
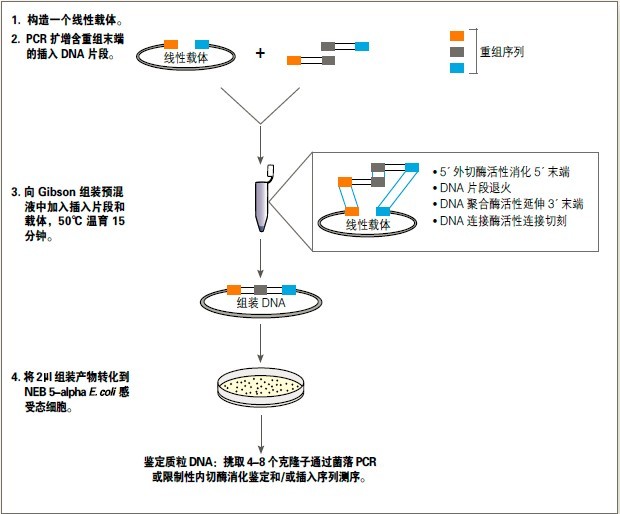
转化到 NEB 5-alpha 高效 E. coli 感受态细胞
• NEB 5-alpha 高效 E. coli 感受态细胞(包含在试剂盒中)建议用于组装< 20 kb 的片段
• 大肠杆菌对有些 DNA 结构有选择拮抗,如倒置和串联重复序列,可能会导致转化效率极低或者生长极差的小
克隆
• 电转化可以把转化效率提高几个数量级,因此 Gibson 组装预混液用于电击转化时,必须把反应体系稀释
3 倍,再取 1 μl 用于转化
下一篇:遗传标记